专注AIGC领域的专业社区,关注微软&OpenAI、百度文心一言、讯飞星火等大语言模型(LLM)的发展和应用落地,聚焦LLM的市场研究和AIGC开发者生态,欢迎关注!
去年6月25日,Evolutionaryscale发布了,拥有980亿参数的全球最大生物模型ESM3。
Evolutionaryscale将蛋白质的三维结构和功能转化为离散的字母表,并构建了一种全新方法,将每个三维结构表示为字母序列,使得ESM3能够同时处理蛋白质的序列、结构和功能,能够响应结合原子层面细节与高层次指令的复杂提示,生成全新的蛋白质。
与自然进化相比,ESM3的模拟进化能力相当于5万亿年。

API地址:https://forge.evolutionaryscale.ai/console
当时这个模型在生物、医药界引起不小的轰动。今天早上4点,Evolutionaryscale宣布正式免费开放ESM3,帮助全球的科学家加速对蛋白质的预测效率。
当得知ESM3开放API后,图灵奖获得者、Meta首席科学家Yann LeCun非常赞赏Evolutionaryscale,认为这是一个非常棒的东西。
作为一名多年来报道AI领域的独立记者,我认为这是一个历史性时刻。ESM3不仅仅是一个模型——它破解了原子级别的蛋白质生成代码。其对医学领域的影响将是惊人的。
ESM3是在全球最强大的GPU集群之一上训练而成,使用了超过1×10^24次FLOPS算力和980亿个参数,是迄今为止在生物模型训练中投入的最大计算资源。
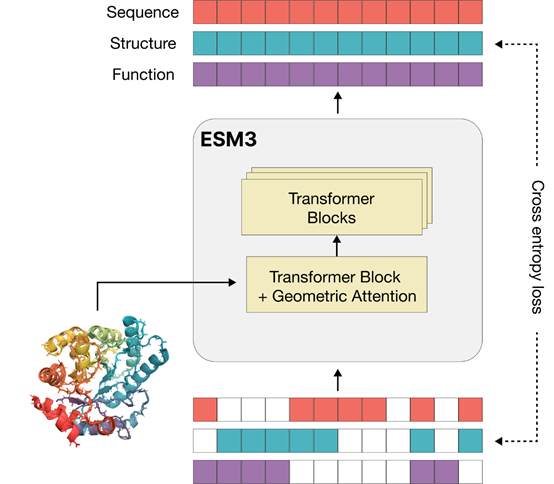
ESM3的核心能力在于它能够同时处理蛋白质的序列、结构和功能,这三种属性对于理解蛋白质的工作原理至关重要。为了实现这一点,ESM3将三维结构和功能转化为离散字母表,使得每个三维结构都可以用一系列字符表示,从而允许模型在大规模上进行训练,并解锁新的生成能力。
这种多模态方法让ESM3可以在处理蛋白质时展现出强大的推理能力,从进化的尺度上学习序列、结构与功能之间的深层次联系。
在训练过程中,ESM3采用了掩码语言建模目标,即对蛋白质的序列、结构和功能进行部分掩盖,然后让模型预测这些掩盖的部分。迫使ESM3深入学习序列、结构和功能之间的联系,从而在数十亿种蛋白质和参数的规模上模拟进化过程。
ESM3的多模态推理能力使其能够以前所未有的精确度生成新蛋白质。例如,科学家可以指导ESM3结合特定的结构、序列和功能要求,生成具有特定活性位点的蛋白质支架。这种能力在蛋白质工程领域具有巨大的应用潜力,尤其是在设计能够分解塑料废物的酶等方面。ESM3的另一个显著特点是其随着规模扩大而出现的能力。
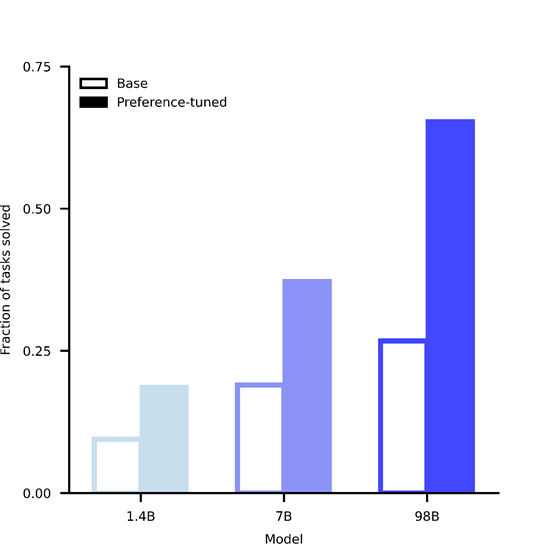
在解决复杂的蛋白质设计任务时,如原子配位任务,ESM3表现出随着模型规模的增大,其解决问题的能力也越强。此外,ESM3还可以通过自我反馈和实验室数据进行自我改进,进一步提升其生成蛋白质的质量。
在实际应用中,ESM3已经展现出了惊人的能力。例如,成功地生成了一种新的绿色荧光蛋白(esmGFP),其序列与已知的荧光蛋白仅有58%的相似度。
通过实验结果显示,esmGFP的亮度与自然界的GFP相似,但其进化路径与自然进化不同,ESM3通过模拟进化过程,相当于在短时间内完成了超过5亿年的自然进化。
(文:AIGC开放社区)


这个ESM3模型真是够猛的,980亿参数还免费开放!多模态能力强大得离谱,科学家们快去研究分解塑料酶吧!