作者:田小幺
编辑:李宝珠
转载请联系本公众号获得授权,并标明来源
美国华盛顿大学 David Baker 团队近期利用先进生成模型,通过合成 OLG 设计研究,从工程化角度验证其可行性。研究团队针对 2 个蛋白家族设计重叠序列,编码高度有序的从头设计蛋白结构,计算机模拟与实验验证均显示出极高成功率。
1977 年,英国生物化学家弗雷德里克·桑格(Frederick Sanger)在解析 ΦX174 噬菌体基因组时,首次发现了一个颠覆认知的现象:这个仅 5.4kb 的 DNA 分子编码的蛋白质总长度,远超其物理容量限制。测序结果揭示,两对基因通过不同阅读框架共享同一 DNA 区域——这种被称为重叠基因(OLG)的现象,在病毒世界中极为普遍。例如,乙型肝炎病毒 3.2kb 基因组中,50% 区域被多对重叠基因覆盖,超过半数已知病毒至少含有一个 OLG。
这种突破直觉的基因组设计,暗藏着病毒的生存智慧:当病毒在宿主细胞内争夺有限空间时,OLG 通过「基因叠罗汉」策略,让单个核苷酸同时参与两个密码子编码,在紧凑序列中实现功能叠加。桑格团队的发现开启了相关研究,后续研究表明,OLG 编码的蛋白质常具有高序列简并性(degenerate),其氨基酸序列容错性使两种功能蛋白能在同一 DNA 链共存。更关键的是,即使需要形成明确三维结构的蛋白质,也能通过序列编排,在不同阅读框架中实现折叠兼容。
然而,核心疑问始终存在:标准遗传密码下,氨基酸序列简并性能否支持任意功能蛋白对在重叠框架中折叠?当核苷酸需兼顾双重编码时,蛋白质折叠的序列空间是否被严重限制?
美国华盛顿大学 David Baker 团队近期利用先进生成模型,通过合成 OLG 设计研究,从工程化角度验证其可行性。研究团队针对两个蛋白家族设计重叠序列,编码高度有序的从头设计蛋白结构,计算机模拟与实验验证均显示出极高成功率:在重叠约束下,可变阅读框架(alternative reading frames)不仅能容纳明确三维折叠,同时其结构稳定性与功能完整性与非重叠序列相当。
相关研究成果以「Design of overlapping genes using deep generative models of protein sequences」为题,已在 bioRxiv 发表预印本。

论文地址:
https://doi.org/10.1101/2025.05.06.652464
关注公众号,后台回复「合成 OLG」获取完整 PDF
开源项目「awesome-ai4s」汇集了百余篇 AI4S 论文解读,并提供海量数据集与工具:
https://github.com/hyperai/awesome-ai4s
数据集:整合多维度数据资源与分析方法
为解析遗传密码可塑性及其在蛋白质设计中的应用,研究整合多维度数据资源与分析方法,构建从理论设计到实验验证的完整研究链条。
在遗传密码随机化研究中,该研究基于氨基酸置换(amino acid permutation)与密码子重排(Codon shufflers)策略,生成 1,000 种替代密码子组合。这一数据集通过明确的算法设计保障了样本多样性与均匀性,为评估密码子重排的功能影响提供了统计学基准。
同时,研究选取 3 个具有代表性的二级结构目标蛋白,构建 9 组成对组合,在控制变量的前提下实现实验条件的标准化,有效连接遗传密码变异与蛋白质结构功能的关联性分析。
在蛋白质结构域序列分析环节,该研究从 Pfam 37.0 数据库提取种子序列,通过随机抽样截取长度 100 氨基酸的子区域,并利用马尔可夫模型生成保留 k-mer 分布的合成蛋白序列。该方法融合生物信息学筛选与统计建模,既保留天然蛋白质的序列特征,又通过引入可控随机变量创建对照样本,为后续分析提供了兼具自然属性与人工设计特征的创新数据集。
在蛋白质语言模型嵌入分析中,研究人员提取 ESM2、ESM3 和 ProstT5 的隐藏层特征,经位置平均后通过 UMAP 算法投影至二维空间。通过精准设定 n_neighbors = 15 等参数,高维序列特征被转化为直观的拓扑图谱,在保留序列相似性结构的同时,为跨模型比较提供了统一的可视化框架,展现了计算生物学与数据可视化的前沿结合。
在实验验证阶段,研究人员对 192 个重叠基因进行克隆重组,生成 384 种框架位移蛋白变体。实验严格控制关键参数:37°C 培养 20 小时确保大肠杆菌表达系统稳定,6M 盐酸胍梯度复性方案保障包涵体蛋白正确折叠。这种从分子设计到纯化表征的全流程量化控制,不仅提升了研究结论的可重复性,更为蛋白质工程提供了标准化实验范式。
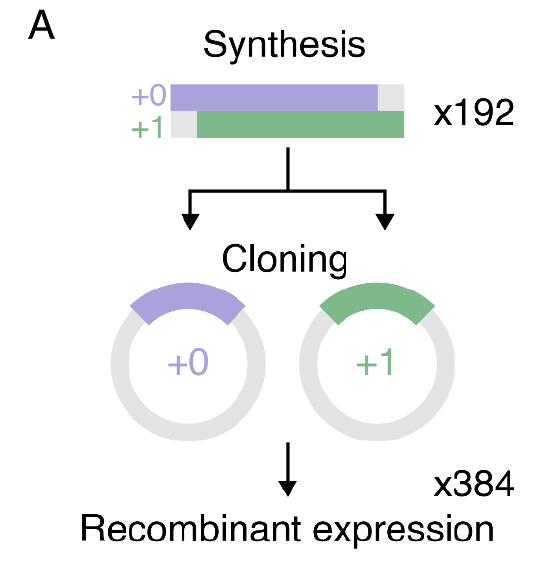
OLG 的克隆重组示意图
基于生成模型的 OLG 设计:多框架兼容的序列同步优化方法
该研究开发了一种计算算法,有效应对重叠基因(OLG)设计中因编码框架相互依赖导致的序列空间受限难题,实现了两个蛋白序列适应性的同步优化。
在算法设计层面,研究整合了 EvoDiff-MSA 和 ProteinMPNN 等生成模型。前者基于 MSA Transformer 架构,通过自回归扩散目标训练,能以目标蛋白多序列比对(MSA)为条件生成设计序列;后者作为结构条件生成模型,可在给定三维结构时设计对应蛋白序列。两类模型均采用逐位置掩蔽与约束采样策略,生成了涵盖多种偏移量和框架排列的重叠序列库。
如下图 A 所示,针对 5 种可变阅读框架(+1、+2、-0、-1、-2)的相位约束,该研究提出逐帧(Frame)迭代采样策略。
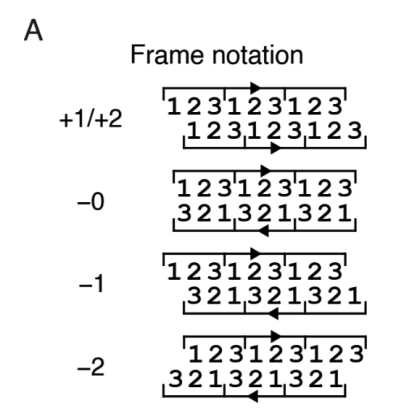
在同一核苷酸序列中编码一对蛋白质的 5 种可能的可变阅读框架
如下图 B 所示,通过分析 -0 框架的氨基酸兼容性矩阵发现,参考框架中单个位置平均存在 2.6 种兼容氨基酸选择,形成 52ⁿ(n 为序列长度)种潜在重叠序列对,凸显遗传密码简并性带来的设计空间。借助蒙特卡洛近似(Monte Carlo approximation)量化其他框架自由度(degrees of freedom),如下图 C 所示,结果显示 +1 和 -1 框架自由度较高(分别约 2.8 和 2.9),而 -2 框架因密码子简并性利用效率低,自由度显著受限(约1.4)。
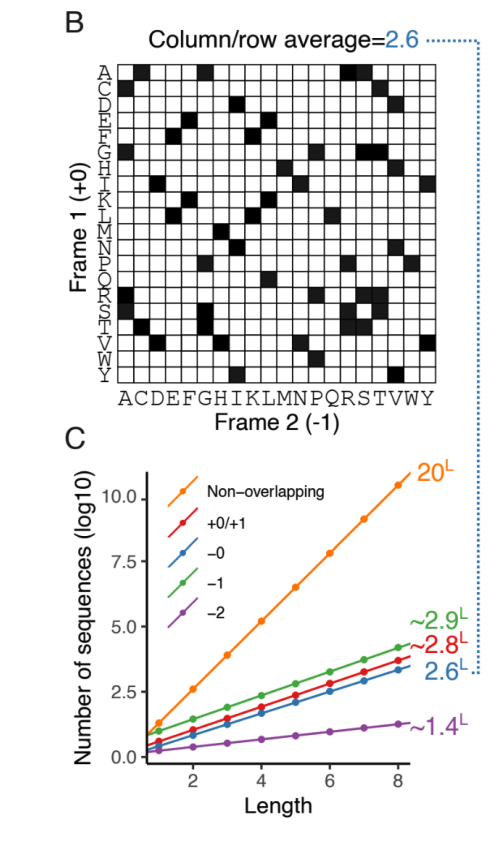
二维兼容性矩阵和蒙特卡洛近似图
最终,如下图 D 所示,算法通过系统扫描序列位置(Scan order),在每次扫描中结合相邻氨基酸约束动态更新联合概率(joint probability)矩阵,经多轮迭代后确保生成的重叠序列对满足框架的兼容性(compatibility)。该策略可扩展至含相位偏移的复杂框架,通过偏置扫描顺序优化设计质量,为生成模型的迭代解码(iterative decoding)提供了关键约束条件。
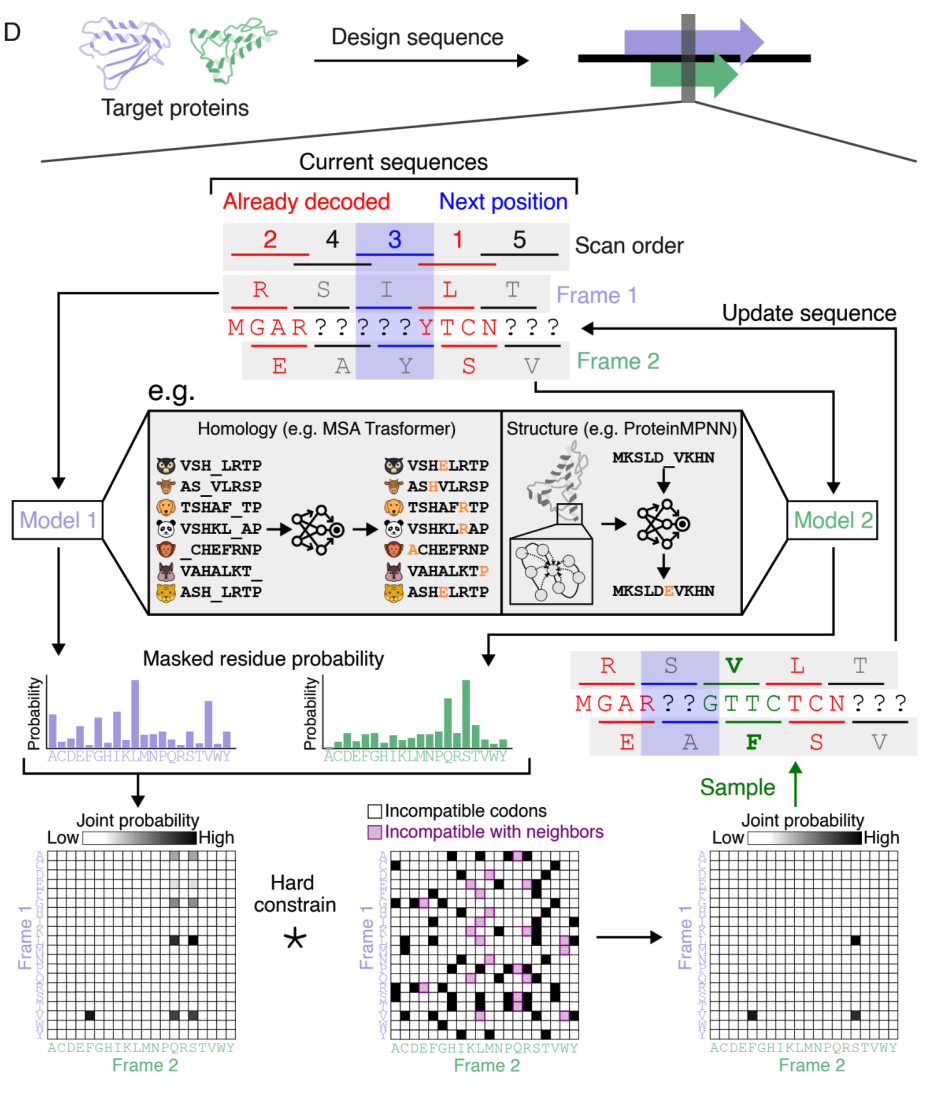
设计 OLG 的约束迭代采样算法示意图
超越天然模板限制:高效生成任意蛋白质对的合成 OLG
实验设计涵盖了多个方向,包括基于同源性的 OLG 设计评估、高度有序蛋白质主链结构的重叠可行性分析、OLG 序列的进化可及性研究以及实验验证。
在基于同源性的 OLG 设计评估中,如下图 A 所示,研究团队选取细菌莽草酸突变酶(CM)与翻译起始因子 1(IF1)为目标,借助 EvoDiff-MSA 生成模型,以多序列比对(MSA)作为条件上下文,经逐位置掩蔽与约束采样,生成 3,307 个完全重叠序列(Overlapping sequence)设计。
如下图 B 所示,尽管设计序列与天然序列同源性仅 38.9%(CM)和 42.3%(IF1),但蛋白质语言模型嵌入分析显示,其在二维空间分布与天然序列高度契合,表明这些设计序列是目标蛋白家族的可信成员,验证了算法对天然蛋白家族的设计能力。
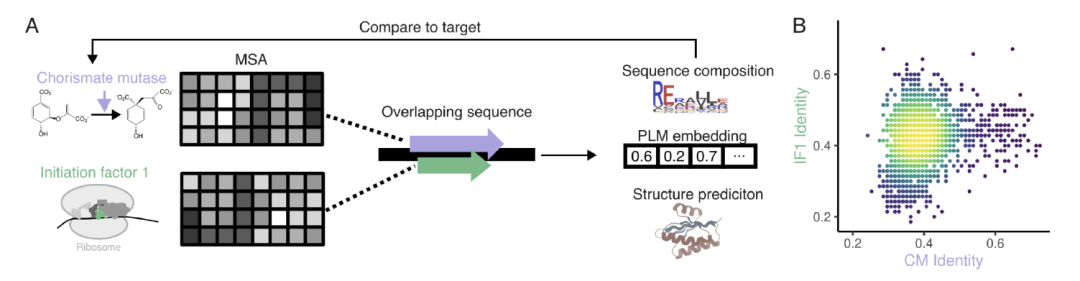
基于同源性的 OLG 设计评估
在探究高度有序蛋白质主链结构的重叠可行性时,如下图 A 所示,研究人员利用 ProteinMPNN 结构条件生成模型,针对 15 种从头生成的主链结构(覆盖 α、β 及混合折叠类别),分别生成 56,250 个重叠设计与 33,000 个非重叠设计。如下图 B 所示,AlphaFold2 评估数据显示,重叠设计的平均 pLDDT 值为 90.2,与非重叠设计的 92.0 接近。
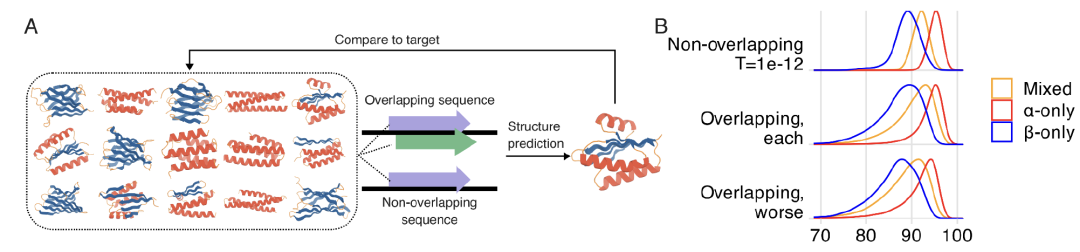
高度有序蛋白质主链结构的 OLG 序列设计分析
进一步分析发现,如下图 C-D 所示,仅 -2 框架因密码子(codons)简并性利用效率低导致表现欠佳。随机化遗传密码分析表明,天然遗传密码(SGC)在编码 OLG 时优势显著,除 -2 框架外均表现良好,且对高简并性氨基酸存在组成偏好,揭示了 SGC 结构对重叠序列可行性的影响机制。
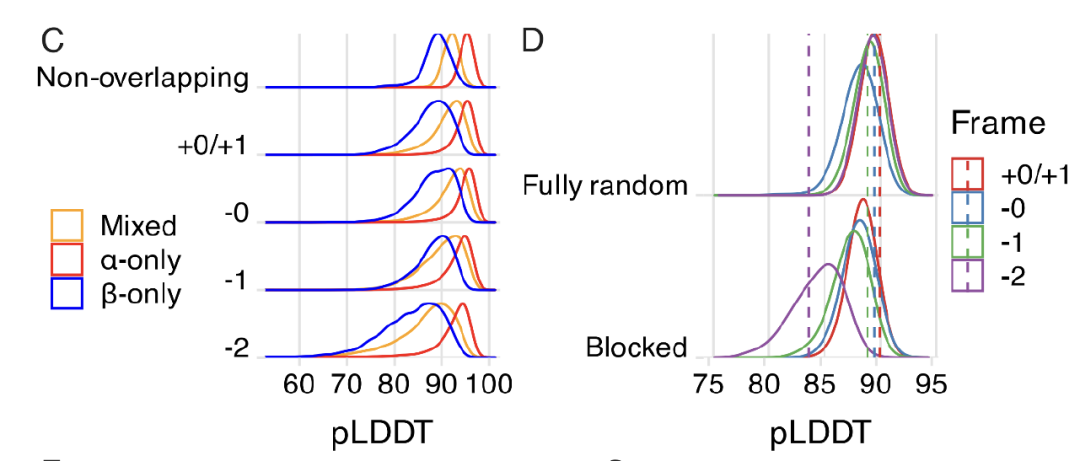
pldts 分布和 AlphaFold2 预测
在进化可及性研究中,研究团队以固定突变数量的种子蛋白序列为起始。如下图 G-F 所示,研究发现即便在零突变的极端条件下,仍有约 1% 的设计能达到高结构稳定性(pLDDT>85,TM>0.7);以天然 Pfam 序列作为亲本时,成功率提升至 3%,且该结果与保留一阶组成偏差的随机序列一致。这充分表明,高度优化的天然蛋白无需大幅序列改变,即可在替代框架中容纳新蛋白,验证了 OLG 在进化层面的可行性。
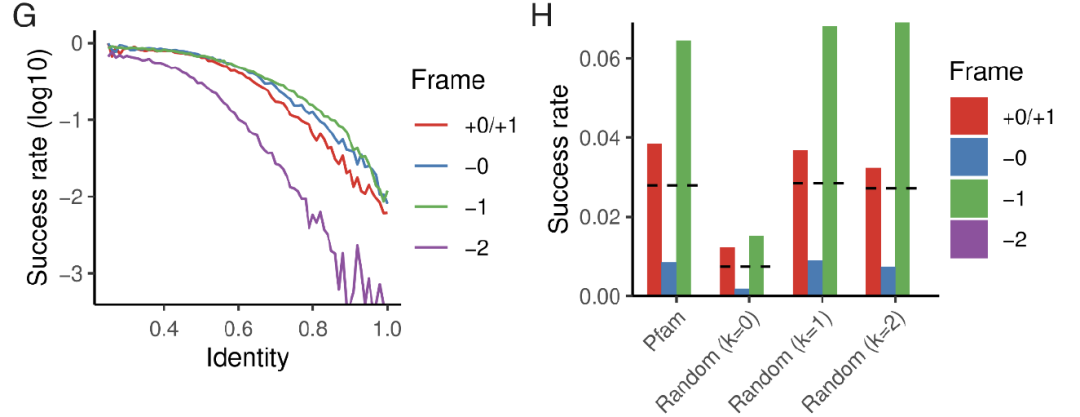
OLG 序列的进化可及性研究结果
最终的实验验证部分,研究团队对 192 个重叠序列进行了重组表达和结构表征。结果表明,如下图 B 所示,54% 的单个蛋白质成功表达,并且大多数具有预期的二级结构和高热稳定性。
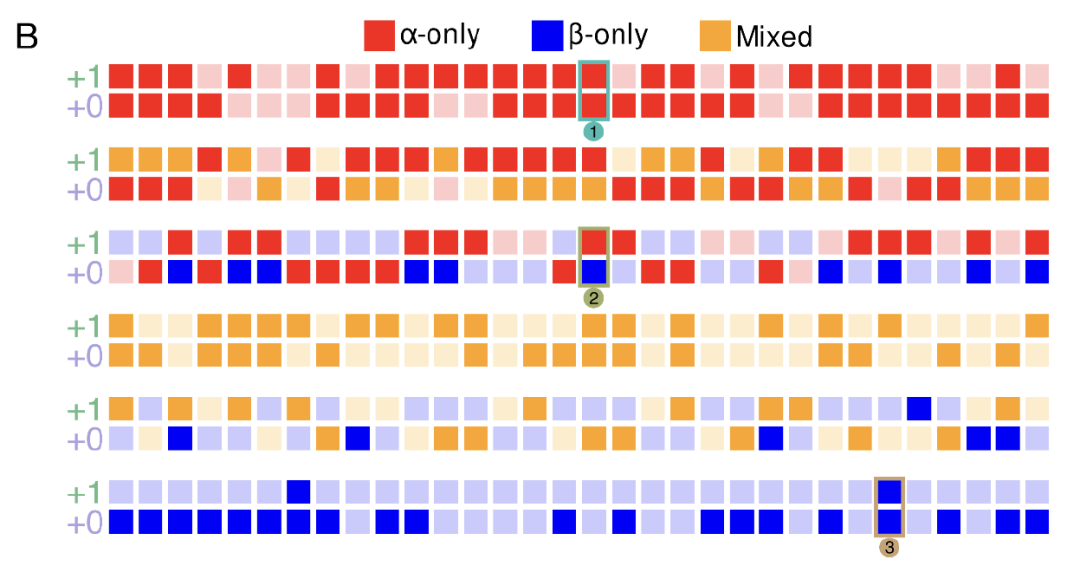
成功表达的蛋白概述
此外,如下图 D-F 所示,成功率因蛋白质的二级结构含量而异,其中 α 螺旋蛋白的成功率最高。此外,31% 的重叠对成功纯化,且一个框架的成功并不影响另一个框架的成功。这些结果进一步支持了 OLG 序列的高可行性和实验验证率,证明了该算法在设计功能性和结构稳定重叠蛋白方面的有效性。

从头设计 OLG 蛋白对的实验表征
合成生物学领域的前沿探索,OLG 工程化应用逐步深入
在合成生物学领域,全球多地的研究团队与企业正投身于对重叠基因(OLG)工程化应用的深入探索之中。
例如,来自清华大学朱听课题组在镜像生物学系统研究方面取得了显著进展,成功实现了全化学合成的镜像 Pfu DNA 聚合酶,这不仅使得千碱基长度的镜像 DNA 组装成为现实,还开发出了基于镜像 DNA 的信息存储技术。这种技术运用镜像基因的编码策略,为 OLG 的双向功能叠加提供了全新的思路。当镜像 DNA 的双螺旋结构同时承载天然与镜像遗传信息时,序列空间的利用率得到了显著提升,为人工基因组的紧凑设计提供了重要的基础。
* 论文链接:https://www.nature.com/articles/s41587-021-00969-6
此外,美国麻省理工学院的 Christopher Voigt 团队开发了基于基因线路设计的合成生物学平台。他们通过重构原核生物基因簇的调控逻辑,成功实现了代谢途径的模块化组装。这种技术路径与 OLG 的设计理念紧密契合。当多个功能基因通过重叠序列形成紧密的遗传模块时,既能减少基因组的冗余,又能通过协同表达提升系统的稳定性。例如,该团队设计的人工固氮基因簇采用 OLG 策略,将多个关键酶的编码序列压缩至同一 DNA 区域,在保证催化效率的前提下,显著降低了宿主细胞的代谢负担。
* 论文链接:https://www.nature.com/articles/s41467-022-33272-2
需要关注的是,这些研究不仅揭示了 OLG 在自然进化中的广泛存在,还通过工程化手段验证了其生物物理上的可行性。在本文所介绍的研究中,David Baker 团队利用深度学习模型设计的合成 OLG,在计算机模拟中展现出了与天然序列相当的结构稳定性。实验验证的高成功率进一步证明了重叠编码的生物学兼容性。这种从基础研究到应用转化的完整闭环,正在重塑合成生物学的设计逻辑,有望在创新药物研发、精准诊断、细胞治疗等多个领域带来全新突破。
参考资料:
1.https://www.tsinghua.edu.cn/info/1181/86148.htm
2.https://tech.huanqiu.com/article/9CaKrnJUV0x
3.https://news.bioon.com/article/4161e88572ad.html


戳“阅读原文”,免费获取海量数据集资源!
(文:HyperAI超神经)