今天,我要和大家探讨一种可能改变脑肿瘤诊断效率的技术——基于向量数据库的智能图像搜索。
脑肿瘤的诊断往往因肿瘤大小和位置的多样性而极具挑战性。通常,专业神经外科医生的精准分析是保障 MRI 报告准确性的关键。然而,在许多发展中国家,缺乏熟练医生和系统化的肿瘤知识,这导致从 MRI 扫描中生成诊断报告的过程既耗时又繁琐。
这是否可以通过技术手段改善呢?答案可能在于自动化的向量数据库搜索系统。通过语义搜索功能,KDB.AI 提供了一种高效的解决方案。它能够根据语义上下文,从脑部扫描数据集中快速检索最相似的图像,即使查询内容与数据库内容并非完全一致。
这种能力,不仅能提升诊断效率,还可能帮助医生更精准地了解患者的病情。接下来,让我们深入探讨这一技术如何革新脑部 MRI 图像搜索的流程和应用。
在本教程中,我们将逐步演示如何将图像数据存储到向量数据库中,并通过预训练的神经网络生成称为向量嵌入的数据结构。我们将使用 KDB.AI 的向量数据库产品,来查找与输入查询图像在向量嵌入上相似的图像。
本教程将涵盖以下内容:加载图像数据,创建图像向量嵌入,将嵌入存储到 KDB.AI,查询 KDB.AI 表,搜索与目标图像相似的图像,以及删除 KDB.AI 数据库和表格。
首先获取数据
### !!! Only run this cell if you need to download the data into your environment, for example in Colab### This downloads image dataimport requestsimport osfrom PIL import Imageimport io!mkdir -p ./data/meningioma_tumor!mkdir -p ./data/glioma_tumor!mkdir -p ./data/no_tumor!mkdir -p ./data/pituitary_tumordef get_github_repo_contents(repo_owner, repo_name, branch, folder_path):# Construct the API URLapi_url = f"https://api.github.com/repos/{repo_owner}/{repo_name}/contents/{folder_path}?ref={branch}"# Send the request and process the responsecontents = requests.get(api_url).json()# Create the local directory if it doesn't existfPath = f"./data/{folder_path.split('/')[-1]}"for item in contents:# Recursively list contents of subfolderif item['type'] == 'dir':get_github_repo_contents(repo_owner, repo_name, branch, f"{folder_path}/{item['name']}")# Download and save fileelif item['type'] == 'file':file_url = f"https://raw.githubusercontent.com/{repo_owner}/{repo_name}/{branch}{folder_path}/{item['name']}"print(file_url)r = requests.get(file_url, timeout=4.0)r.raise_for_status() # Raises an exception for HTTP errorswith Image.open(io.BytesIO(r.content)) as im:im.save(f"{fPath}/{item['name']}")# Get dataget_github_repo_contents(repo_owner='KxSystems',repo_name='kdbai-samples',branch='main',folder_path='/image_search/data')

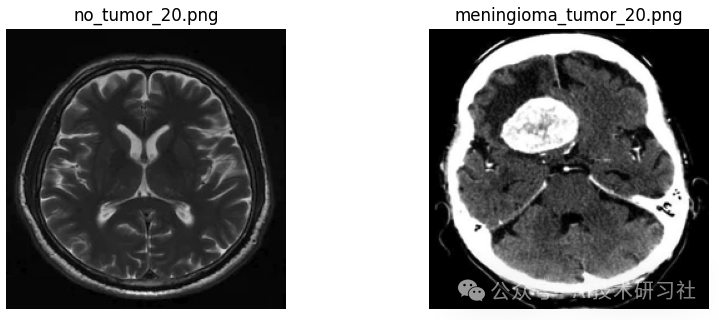
此示例中使用的数据集是从 Kaggle 获取的脑肿瘤分类图像数据集。该数据集由 MRI 脑部扫描图像组成,根据图像中是否存在肿瘤分为四类:神经胶质瘤、黑脑膜瘤、垂体瘤和无肿瘤。
原始 Kaggle 数据集包含两个文件夹:Training 文件夹和 Testing 文件夹,它们均按照上述肿瘤类别对图像进行分类和组织。作为预处理步骤,我们已将原始图像的大小调整为 (224, 224, 3),其中高度和宽度均为 224,红色、绿色和蓝色像素强度分别占据 3 个通道。此外,我们重新命名了每个图像文件,使其名称对应于类别,并在其目录中为每个图像分配了唯一的 ID。
在数据后处理阶段,我们将原始数据集中的 Training 文件夹用于训练 ResNet 模型,该模型将在本教程中用于生成向量嵌入。同时,经过后处理的 Testing 文件夹已重命名为 data,并将在本教程中使用。需要注意的是,这些数据并未被 ResNet 模型见过,因此在生成向量嵌入时可以有效避免过拟合问题。
接下来,让我们从 ‘Testing’ 目录中的不同子文件夹中提取图像文件路径。这些需要传递给下一节中的函数以创建嵌入。
def extract_file_paths_from_folder(parent_dir: str) -> dict:image_paths = {}for sub_folder in os.listdir(parent_dir):sub_dir = os.path.join(parent_dir, sub_folder)image_paths[sub_folder] = [os.path.join(sub_dir, file) for file in os.listdir(sub_dir)]return image_paths
image_paths_map = extract_file_paths_from_folder("data")
该 image_dataset_from_directory() 函数使用与图像目录对应的类标签保存每个图像。这是一种快速简便的方法,可以将我们的数据及其标签以正确的格式进行嵌入。
dataset = image_dataset_from_directory("data",labels="inferred",label_mode="categorical",shuffle=False,seed=1,image_size=(224, 224),batch_size=1,)
为了创建图像嵌入,我们将使用一个已经在脑肿瘤分类问题上预先训练过的神经网络。在此示例中,我们采用包含 ResNet-50 主干网络的模型。ResNet-50 是一种常用于图像分类任务的流行神经网络架构,具有良好的通用性能。
ResNet-50 最初是在 ImageNet 数据集上训练的——尽管 ImageNet 数据集包含数百万张图像,其中包括 MRI 扫描图像,但并未涵盖不同类型脑肿瘤的示例。因此,我们的自定义模型是在 ResNet-50 的基础上构建的:首先在 ImageNet 上进行预训练,然后重新训练以适应 MRI 脑部扫描图像分类的特定需求。
这实际上是迁移学习的一个典型应用——我们利用一个已为一般任务(ImageNet 图像分类)预训练的模型(ResNet-50),并将其作为解决更具体问题(MRI 脑部扫描图像分类)的起点。迁移学习的优势在于可以显著减少训练时间,同时充分利用预训练模型中已有的特征学习能力,从而提高特定任务的表现。
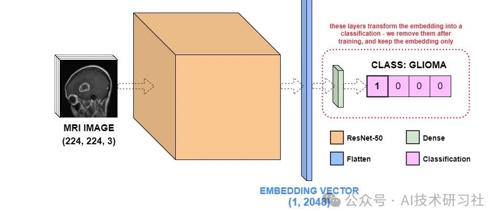
model = from_pretrained_keras("KxSystems/mri_resnet_model")该模型有四个层:ResNet-50、Flat 和两个 Dense 层。ResNet-50 “层” 实际上是许多层,抽象在一个名称下。这就是它包含数百万个参数的原因。Flatten 层不包含任何参数 – 其唯一目的是将 ResNet-50 的输出“展平”为 2048 维向量,称为特征向量。最后两个 Dense 层将 ResNet-50 特征向量转换为输入图像的 4 维分类。
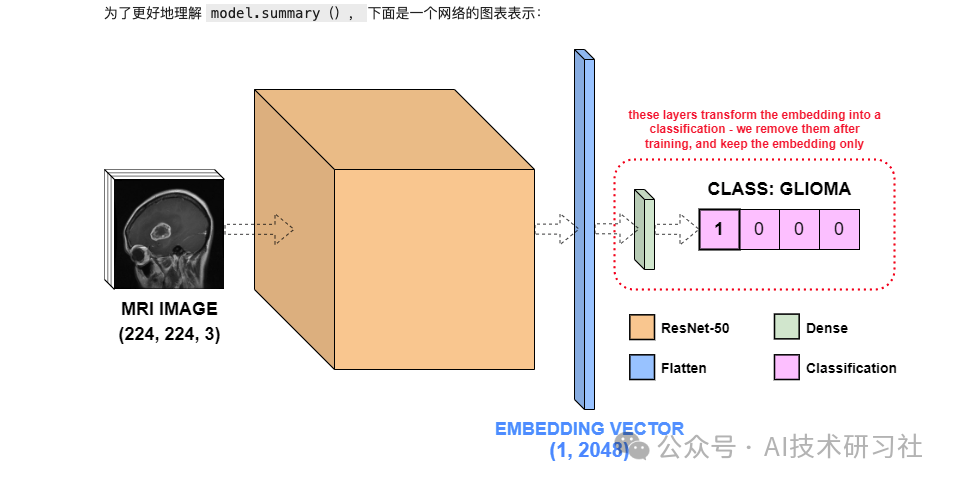
尽管 Dense 层对于训练 ResNet-50 对我们的四种脑肿瘤类别进行分类至关重要,但我们将不再需要它们。在此示例中,我们感兴趣的是嵌入,而不是分类输出。因此,我们将通过调用 pop() 来删除预训练模型的最后两层。这意味着模型的新输出是 2048 维特征向量 – 输入图像的 ResNet-50 嵌入。
为了生成图像嵌入,我们将遍历数据集,通过在图像上调用 model.predict() 来获取 2048 维嵌入,并保存相应的类向量。
# create empty arrays to store the embeddings and labelsembeddings = np.empty([len(dataset), 2048])labels = np.empty([len(dataset), 4])
# for each image in dataset, get its embedding and class labelfor i, image in tqdm(enumerate(dataset), total=len(dataset)):embeddings[i, :] = model.predict(image[0], verbose=0)labels[i, :] = image[1]
创建嵌入后,我们需要将它们存储在向量数据库中以实现高效搜索。
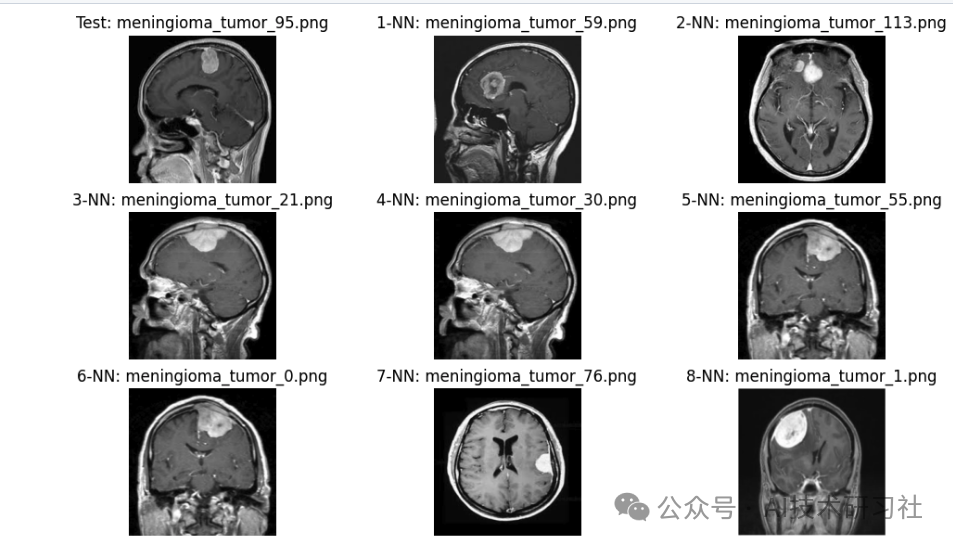
我们可以看到,通过第二个测试图像,我们得到了一组相似的大脑扫描,它们与第二个测试图像非常匹配,并且都属于同一类别。这种结果可以帮助医生确认他们自己的假设。
完整代码参考:https://github.com/KxSystems/kdbai-samples/blob/main/image_search/image_search.ipynb
(文:AI技术研习社)